2位作者,发了一篇Nature大子刊:开发细菌脱氨酶驱动的RNA编辑平台!
时间:2024-07-07 23:04:30 热度:37.1℃ 作者:网络
RNA编辑是一种广泛存在于真核生物及其相关病毒中的自然过程,通过插入、删除或转换核碱基,使RNA序列与编码基因不同。最常见的RNA编辑类型是腺苷(A)到次黄嘌呤(I)的转换,由作用于RNA的腺苷脱氨酶(ADARs)介导。
相比于通过修改细胞基因组来改变细胞命运的DNA编辑,RNA编辑可以在不引入永久性遗传改变的情况下改变细胞行为,具有可逆性和剂量依赖性,特别适合需要特定编辑水平或治疗时间窗口的疾病治疗。现有的程序性RNA编辑技术主要依赖于ADARs,但这些系统存在一定的局限性,例如在大多数成人组织中表达量低以及脱靶效应较高。
2024年7月5日,芝加哥大学的汤玮欣教授(通讯作者)和其课题组成员Hao Yan博士后(第一作者)两人在Nature Chemical Biology期刊上发表了一篇名为:Programmed RNA editing with an evolved bacterial adenosine deaminase的论文。
研究人员开发了名为DECOR(细菌脱氨酶驱动的RNA重编码)的系统,利用进化的大肠杆菌转移RNA腺苷脱氨酶(TadA8e),在CRISPR指定的人类转录组位点上实现腺苷到次黄嘌呤的编辑。DECOR能够在多种细胞类型中工作,包括人类肺成纤维细胞,并且在目标位点上的活性与过表达ADAR的RNA编辑平台相似,但脱靶效应降低了88%。高保真度的DECOR系统将脱靶效应进一步降低到基础水平。通过靶向导致范德伍德综合征的干扰素调节因子6(IRF6)不足,研究展示了DECOR的临床潜力。DECOR介导的RNA编辑移除了IRF6 5'非翻译区中的致病上游开放阅读框(uORF),使主要ORF表达从12.3%恢复到36.5%,接近健康转录本水平。DECOR拓展了现有效应蛋白的种类,为程序性RNA编辑开辟了新领域。

一种新的RNA编辑平台DECOR(细菌脱氨酶驱动的RNA重编码)
首先,研究人员筛选了几种可能用于RNA编辑的效应蛋白,包括能够作用于DNA和RNA的胞嘧啶脱氨酶rAPOBEC1、高活性去氨胞嘧啶的APOBEC家族蛋白hA3A、进化的脱氧胞苷脱氨酶evoCDA1、大肠杆菌tRNA腺苷脱氨酶TadA及其衍生物TadA7.10、TadA8.20和TadA8e。为了实现编程能力,他们选择了来自Ruminococcus flavefaciens的RNA靶向CRISPR系统RfxCas13d,并将效应蛋白通过柔性连接子与核酸酶缺陷型dCas13d融合。
通过瞬时转染将表达dCas13d融合蛋白和编码gRNA的质粒引入人类胚胎肾(HEK)293T细胞,并对EZH2 mRNA进行逆转录、PCR扩增和下一代测序(NGS)分析。他们发现,尽管某些效应蛋白如hA3A没有显著的编辑效果,但TadA7.10、TadA8.20和TadA8e表现出明显的编辑活性,其中TadA8e的编辑效率最高。
研究还表明,DECOR平台与多种CRISPR系统(包括LwaCas13a、PspCas13b和Cas13X.1)兼容,尽管RfxCas13d系统表现出最强的RNA编辑信号。进一步的实验表明,DECOR能够在多种目标基因上实现RNA编辑,其中RfxCas13d仍然是最有效的系统。
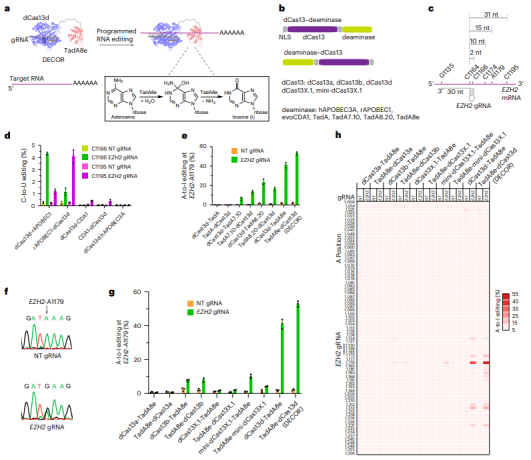
图1:通过dCas13–脱氨酶融合实现的程序性RNA编辑。
DECOR在人体细胞中实现高效RNA编辑的能力
首先,研究人员选择了七个目标位点,并设计了DECOR和REPAIR系统以编辑相同的A位点。在比较实验中,DECOR和REPAIRv1都展示了最低的A-to-I转换率(0.8-1.8%),但在特定gRNA的存在下,这一转换率显著提高,DECOR达到了19.7-52.9%的编辑效率,而REPAIRv1为0.6-63.5%。此外,DECOR在处理长非编码RNA MALAT1时表现尤为出色,达到39.8%的编辑率,而REPAIRv1的效率较低。这表明DECOR和REPAIRv1具有功能互补性。
研究还发现,DECOR在不同的CRISPR系统中兼容性良好,尽管RfxCas13d系统表现最优。通过进一步实验,研究人员在HeLa(宫颈癌细胞系)和A549(肺癌细胞系)中验证了DECOR的编辑效率,并在原代细胞中(如人肺成纤维细胞)中也表现出较高的编辑水平。
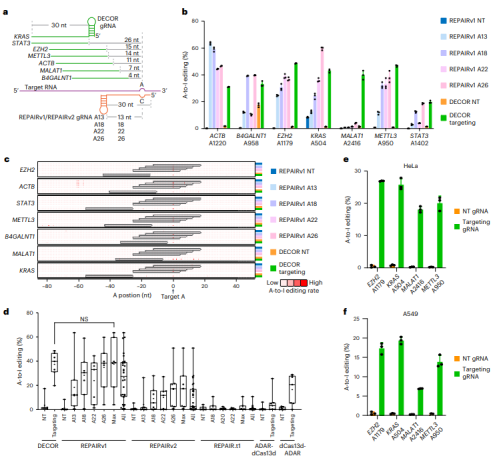
图2:DECOR介导的编码和非编码RNA编辑。
DECOR在全转录组范围内的脱靶效应
首先,研究人员在HEK293T细胞中使用EZH2靶向和非靶向gRNA对五个RNA编辑平台进行了脱靶效应检测,结果显示与之前的观察一致。为了全面评估DECOR的脱靶行为,还使用了KRAS和MALAT1的两个额外gRNA。
结果表明,REPAIRv1在使用非靶向和EZH2靶向gRNA时检测到4,044和4,004个脱靶位点,编辑水平中位数分别为30.6%和29.8%。许多脱靶编辑位点似乎是gRNA非依赖性的,因为非靶向和EZH2靶向REPAIRv1之间共享了2,426个脱靶位点。
相比之下,REPAIRv2、REPAIR.t1和dCas13d-ADARDD-E488Q显著减少了脱靶位点的数量。REPAIRv1的嵌入高活性ADAR变体显示在人类细胞中具有相当大的全转录组脱靶效应,而DECOR在使用非靶向和EZH2靶向gRNA时分别检测到888和840个脱靶位点,比REPAIRv1减少了约88%。DECOR的脱靶位点的中位编辑水平也显著低于REPAIRv1。
此外,DECOR和REPAIRv1的脱靶位点几乎完全不同,这表明脱氨酶而不是CRISPR复合物决定了这些RNA编辑系统与人类转录组的相互作用。DECOR的脱靶编辑位点富集于YA序列,UA最为偏好,而REPAIRv1则偏好于G和C在目标A之后的序列。
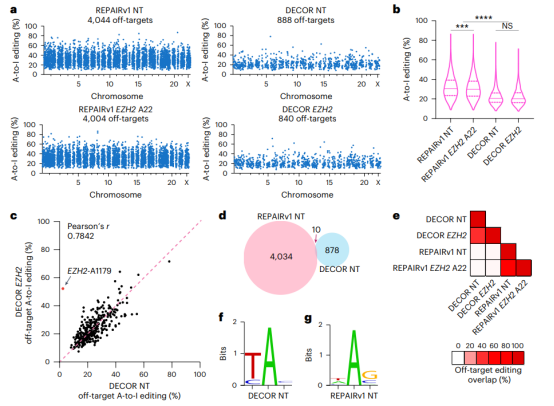
图3:DECOR在转录组范围内的特异性。
高保真DECOR降低脱靶编辑效应
研究人员引入了F148A和V106G两个突变来降低DECOR的脱靶效应,并命名为hfDECOR。hfDECOR-F148A和hfDECOR-V106G在18个内源性RNA中分别产生了23.1 ± 10.5%和24.0 ± 9.2%的A-to-I编辑,略低于DECOR的35.4 ± 12.1%。hfDECOR在非靶向gRNA的情况下显示出显著减少的脱靶效应,hfDECOR-F148A和hfDECOR-V106G分别检测到47和126个脱靶位点,比DECOR分别降低了94.7%和85.8%。hfDECOR的脱靶编辑位点的中位编辑水平也显著低于DECOR。结果表明,hfDECOR提供了稳健且干净的RNA编辑。
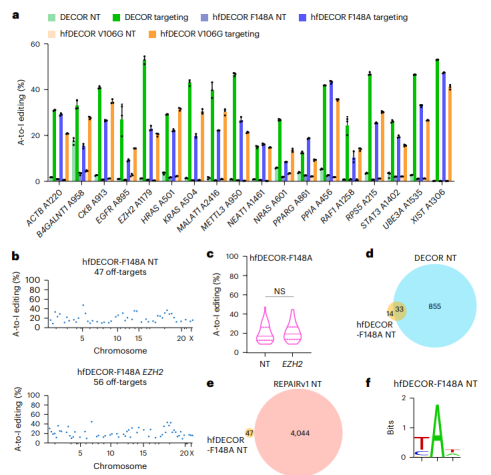
图4:高保真DECOR的效率和特异性。
DECOR修复早期终止密码子并恢复翻译
研究人员尝试利用DECOR重新编码含有早期终止密码子的绿色荧光蛋白(GFP)mRNA。设计了四个gRNA,将UAG密码子放置在原核RNA探针的2–32 nt下游。其中,gRNA b在UAG密码子上游11–40 nt处结合,导致GFP荧光的强恢复。高通量测序确认UAG密码子的编辑水平为7.0%,证明观察到的GFP恢复确实是RNA编辑的结果(图5a-c)。
DECOR去除导致疾病的uORF以表达pORF
研究人员探索了DECOR在去除IRF6 5′ UTR中导致疾病的uORF方面的潜在临床应用。Van der Woude综合症(VWS)的一些患者携带A–48T突变,该突变在IRF6的5′ UTR中创建了一个新的起始密码子,抑制了IRF6蛋白的正常表达。研究人员提出编辑A–49以静默uORF,将疾病相关的起始密码子AUG转换为IUG。使用双荧光素酶报告基因检测确认A–48T突变显著降低了pORF的表达。将A–49突变为G在A–48T背景下显著提高了pORF的表达。高通量测序确认A–49的编辑水平为10.2%,RNA编辑将表达水平提高到健康UTR的36.5%(图5d-i)。
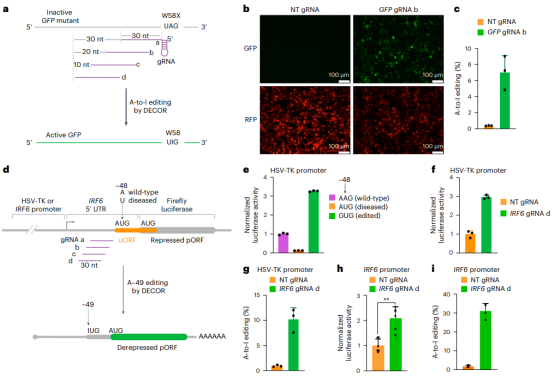
图5:通过DECOR移除致病性上游开放阅读框(uORF)并恢复蛋白质表达。
【小结】
总之,这篇论文主要介绍了DECOR作为一种新型的CRISPR基础RNA编辑平台的开发和特性。DECOR利用进化的细菌tRNA腺苷脱氨酶,不依赖于ADAR,能在CRISPR引导下实现A到I的编辑,其编辑效率与传统ADAR介导的RNA编辑相似,但具有显著较低的全转录组脱靶效应。通过高保真版本,DECOR能进一步将脱靶效应降至接近基础水平。此外,DECOR展示了在编码序列和UTR等多个编辑点需求的场景中的应用潜力,尤其在早期发育阶段调节转录本功能可能具有重要临床应用前景。
原文链接:
https://doi.org/10.1038/s41589-024-01661-x


